제2회 AI 신약개발 경진대회, 우수상 후기


제 2회 AI 신약 개발 경진 대회: 도전
안녕하세요, 히츠 AI 연구팀의 배성한입니다.
지난 7월, 한국제약바이오협회 주관으로 제2회 AI 신약개발 경진대회가 열렸습니다. 작년 1회 대회는 제가 히츠에 합류한 후 맡은 첫 프로젝트였는데요. 그때는 저희 AI 연구팀과 함께 팀을 이루어 참여했었습니다. 팀원들과의 긴밀한 토론과 협력을 통해 순위를 서서히 올려, 700여 개 팀 중 최종 4위를 달성한 좋은 기억이 있습니다.

지난 1년 동안 제가 히츠에서 얼마나 성장했는지를 스스로 시험해보고자, 올해는 혼자 대회에 참여하게 되었습니다. 그리고 이번 대회에서도 4위라는 성적으로 우수상을 수상하게 되어, 지난 10월 31일에 시상식이 열린 AI Pharma Korea 2024 컨퍼런스에 참석하게 되었습니다. 이번 포스팅에서는 제가 이번 대회에서 시도한 여러 AI 기반 신약 개발 기술들을 소개하고, 수상 후기에 대해서도 짤막하게 공유하고자 합니다.

데이터 부족, 어떻게 극복할 수 있을까?
이번 제2회 AI 신약개발 경진대회의 과제는, 면역 반응 신호 전달에 관여하는 단백질 카이네이즈(kinase)의 일종인 IRAK4를 대상으로 한 약물의 억제 능력을 나타내는 IC50 값을 예측하는 AI 모델을 구축하는 것이었습니다.
총 1,952종의 약물에 대한 IC50 값만이 학습 데이터로 제공되었는데, 이는 머신러닝 학습에 사용되기에는 매우 적은 양으로, 작년 대회와 마찬가지로 이번 경진대회의 난이도를 높이는 주요 요인이 되었습니다. 적은 데이터로 학습된 AI 모델은 학습에 사용된 데이터의 패턴만 기억하게 되어, 학습 데이터 외의 테스트 케이스들에 대해서는 잘 예측하지 못하는 현상인 ‘과적합(Over-fitting)’이 발생할 가능성이 크기 때문입니다.
따라서 이번 대회의 핵심은 데이터 부족을 어떻게 극복하느냐에 있었다고 할 수 있습니다. 저는 아래 세 가지 주요 전략을 활용하여 이 문제를 해결하고자 했습니다.
-
GNN(Graph Neural Network) 형태의 사전학습 모델인 MolCLR 기반 모델 구축
-
IC50 라벨 분포의 불균형을 완화하기 위한 LDS(Label Distribution Smoothing)와 FDS(Feature Distribution Smoothing) 기법 활용
-
IC50 예측에 힌트가 되는 Binding affinity 예측 값 활용
분자 데이터를 학습하다: MolCLR의 활용
먼저, 이번 경진대회에서 제 AI 모델의 backbone이 되는 MolCLR에 대해 소개하고자 합니다. MolCLR는 방대한 양의 분자 데이터를 그래프 형태로 사전 학습(Pre-training)한 GNN 모델입니다. 사전 학습이란, 예측 목표에 대한 본격적인 학습 이전에 해당 예측 목표와 연관된 도메인의 데이터를 AI 모델에 미리 학습시켜 놓는 방법입니다.
이러한 사전 학습을 통해 AI 모델은 해당 도메인에 대한 일반적인 지식을 먼저 습득하게 되고, 이를 바탕으로 상대적으로 적은 데이터만으로도 과적합 없이 효과적으로 학습할 수 있습니다. 특히 컴퓨터를 통해 얼마든지 생성할 수 있는 이미지나 텍스트 데이터와는 달리, 실험을 통해서만 얻을 수 있는 과학적 데이터는 매우 한정적이기 때문에, 사전 학습 모델의 구축과 활용이 과학 분야에서도 활발히 이루어지고 있습니다.
그중 대표적인 분자 구조 사전 학습 모델 중 하나인 MolCLR은 세계 최대 화학 데이터베이스인 PubChem에 속한 천만 개 이상의 분자를 미리 사전 학습한 모델입니다. MolCLR은 PubChem에서 가져온 실제 분자와, 원자나 결합 또는 작용기를 무작위로 가리거나 제거하여 만든 가짜 분자를 구분하는 대조 학습(Contrastive Learning)을 통해, 분자가 지닌 구조적·화학적 특성을 학습합니다. 이렇게 AI 모델이 습득한 사전 지식은, 학습 데이터가 가지는 부족한 구조적·화학적 다양성을 보완해 줌으로써 학습 과정에서 과적합을 방지하는 데 효과가 있습니다.
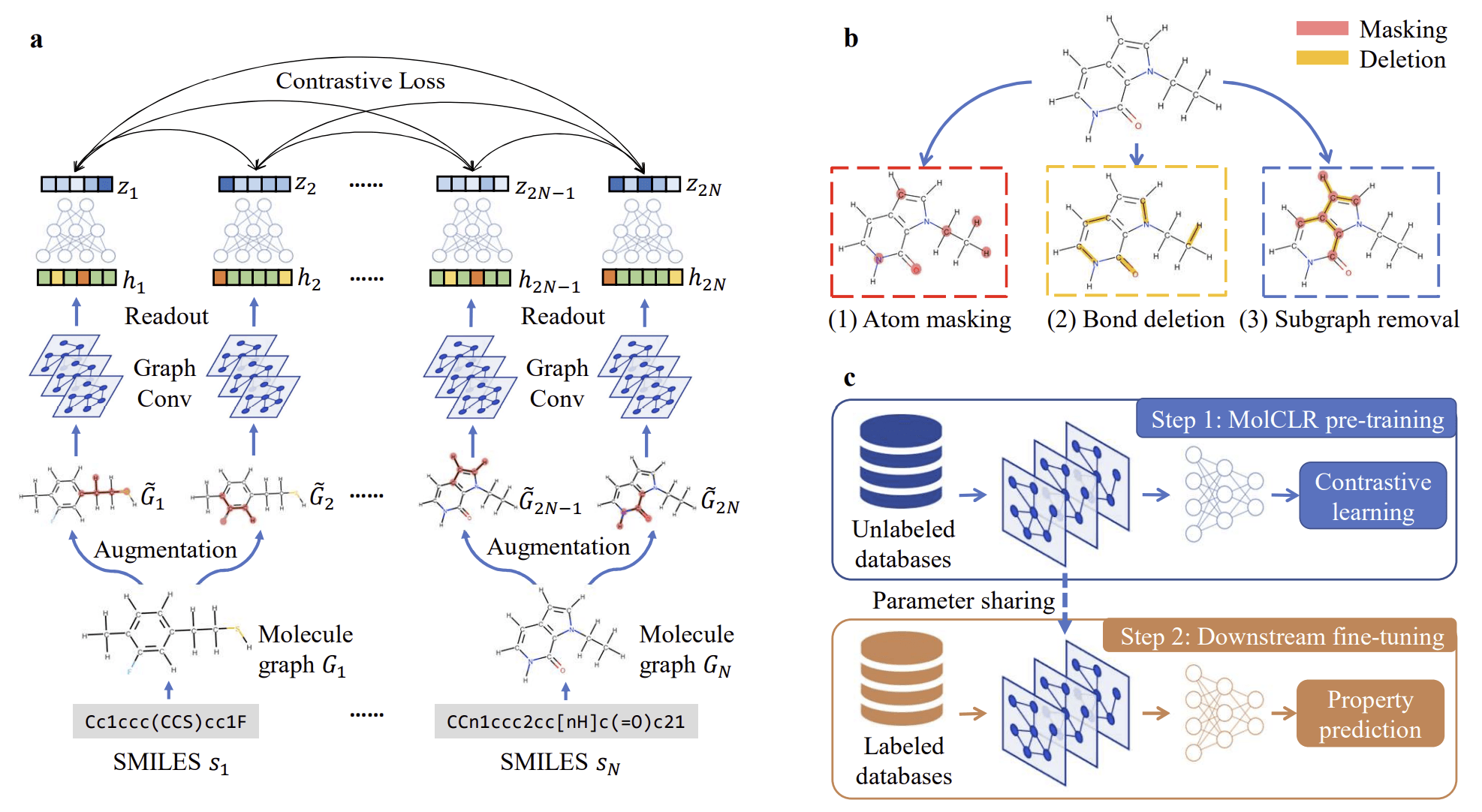
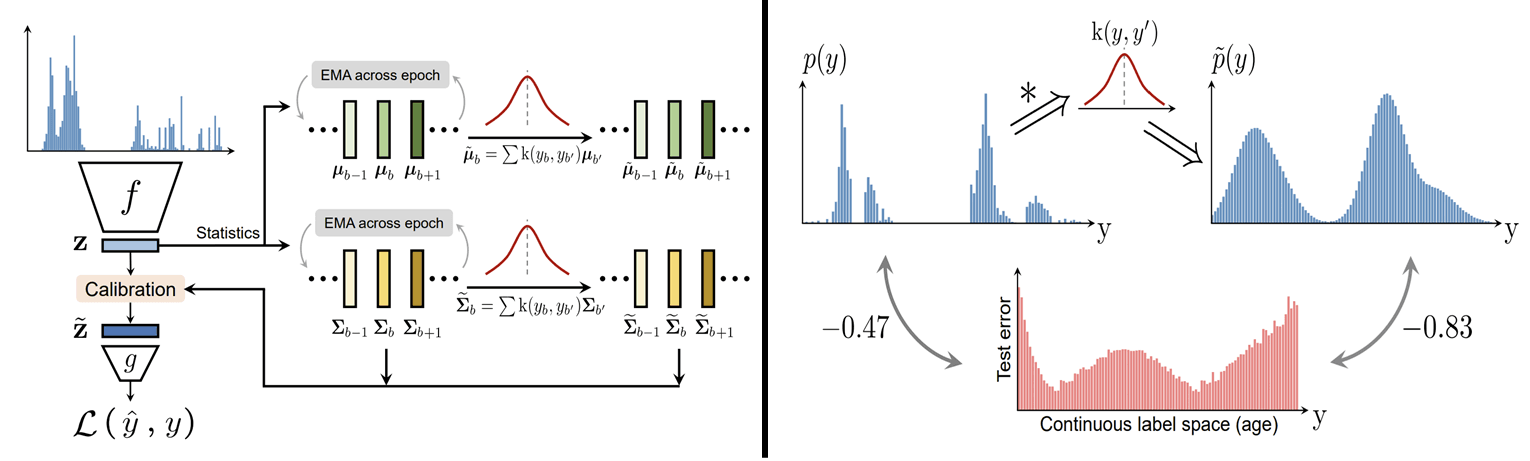
과적합을 방지하기 위해 활용한 또 다른 전략은, 학습 데이터의 불균형한 라벨(IC50) 분포를 완화하는 것이었습니다. 라벨의 불균형은 학습 데이터 자체의 다양성만큼이나 모델의 과적합에 큰 영향을 미칩니다. 아무리 구조적·화학적으로 다양한 데이터를 확보한다 하더라도, IC50 분포가 낮은 쪽으로 치우쳐 있다면 해당 데이터로 학습된 AI 모델은 높은 IC50 값을 제대로 예측하지 못할 수 있습니다.
이러한 라벨의 불균형으로 인한 과적합을 방지하기 위해, 모델이 학습 과정에서 보지 못한 라벨도 예측할 수 있도록 보완해 주는 LDS(Label Distribution Smoothing)와 FDS(Feature Distribution Smoothing) 기법을 도입하였습니다.
LDS와 FDS로 불균형을 넘어서다
LDS와 FDS는 2021년에 발표된 Delving into Deep Imbalanced Regression 논문에서 제안된 딥러닝 모델 학습 기법으로, 연속적인 라벨 값을 예측하는 회귀(Regression) 문제에서 라벨 분포가 불균형할 경우 이를 보정함으로써 AI 모델이 편향된 값을 예측하는 것을 방지해 주는 방법입니다. 이름 그대로, 두 방법 모두 학습 데이터의 라벨 분포에 따라 데이터를 매끈하게(Smoothing) 처리해 주는 방식인데요. 그 대상이 라벨 자체인 경우에는 LDS, 딥러닝 모델이 학습하는 데이터의 특징(feature)인 경우에는 FDS라고 합니다.
LDS와 FDS의 개념에 대해 간략하게 설명드리겠습니다. 불균형한 학습 데이터의 분포를 조정하여 부드럽게 이어지는 분포로 근사한 뒤, 모델이 학습하는 feature와 근사된 라벨 분포가 유사하다는 가정 하에 이를 보정해 주는 방식이라고 할 수 있습니다. LDS와 FDS를 도입함으로써 라벨 불균형으로 인한 과적합이 완화되었고, 학습 과정에서의 예측 성능과 테스트에서의 예측 성능 간의 차이가 크게 줄어들었습니다. 이는 제가 대회 기간 중 경험한 두 번의 점프업 중 하나였습니다.
또 다른 점프업은 바로 AI 모델에 inductive bias를 도입한 것이었습니다. Inductive bias란, AI 모델이 특정 태스크를 학습할 때 해당 태스크의 이해에 도움이 될 수 있도록 사전에 주어지는 특수한 가정을 의미합니다. 가장 대표적인 예가 앞서 언급한 MolCLR 모델의 구조인 GNN입니다. GNN은 노드(node)와 엣지(edge)로 구성된 그래프 구조를 모사하여 모델 구조를 구성함으로써, 그래프 형태의 데이터 패턴을 잘 학습할 수 있도록 inductive bias가 적용된 딥러닝 모델입니다. 이러한 inductive bias가 효과적으로 작동하기 때문에, GNN은 그래프 형태의 분자 데이터 학습에 매우 적합한 모델로 여겨지며 활발히 활용되고 있습니다.
Binding Affinity로 IC50의 실마리를 찾다
이번 경진대회 때 모델에 적용한 inductive bias는 binding affinity 예측 값을 모델이 활용할 feature로 제공하는 것이었습니다. 이번 태스크의 목적인 IC50의 개념에 대해 생각해 봅시다. IC50이란 특정 표적 단백질(이번 태스크에서는 IRAK4)의 활성을 50%로 억제하는 데 필요한 약물 농도를 의미합니다. 즉, IC50은 해당 표적 단백질이 속해 있는 시스템(예: 세포나 조직) 전체에 약물을 투여하였을 때의 반응을 정량적으로 측정한 값으로, 거시 단위에서 약물의 억제 능력을 나타내는 척도라고 할 수 있습니다.
하지만 좀 더 미시세계로 들어가면, 약물이 표적 단백질을 억제하는 기작은 해당 약물의 분자가 표적 단백질 내의 특정 위치에 화학적 작용으로 인해 결합함으로써 이루어지는데요. 이때 약물이 단백질에 결합하는 정도를 binding affinity라고 합니다. 즉, 우리는 미시세계에서 약물-단백질 간의 결합력인 binding affinity와, 거시세계에서 억제 효과의 척도인 IC50이 매우 밀접한 관련이 있을 것이라고 추측할 수 있습니다. binding affinity가 IC50 예측 태스크에 힌트가 되는 일종의 inductive bias인 셈입니다. 원래라면 binding affinity 에너지 그 자체도 실험을 통해 측정해야 하는 값이기 때문에, 이를 AI 모델의 학습용 feature로 바로 사용하는 것은 어려운 일입니다.
Hyper Lab, 도약의 기반이 되다
하지만 저희 HITS에서 제공하는 AI 신약개발 플랫폼 Hyper Lab을 이용하면, 표적 단백질과 후보 약물의 구조만 있다면 AI가 예측해 주는 약물-단백질 결합 구조와 binding affinity를 손쉽게 계산할 수 있습니다. 히츠의 단백질-약물 결합 예측 AI 모델은 실제 미시세계에서 단백질과 약물 분자 간의 상호작용을 모사한 네트워크 구조와 알고리즘을 바탕으로 설계되어, 매우 높은 정확도를 자랑합니다.
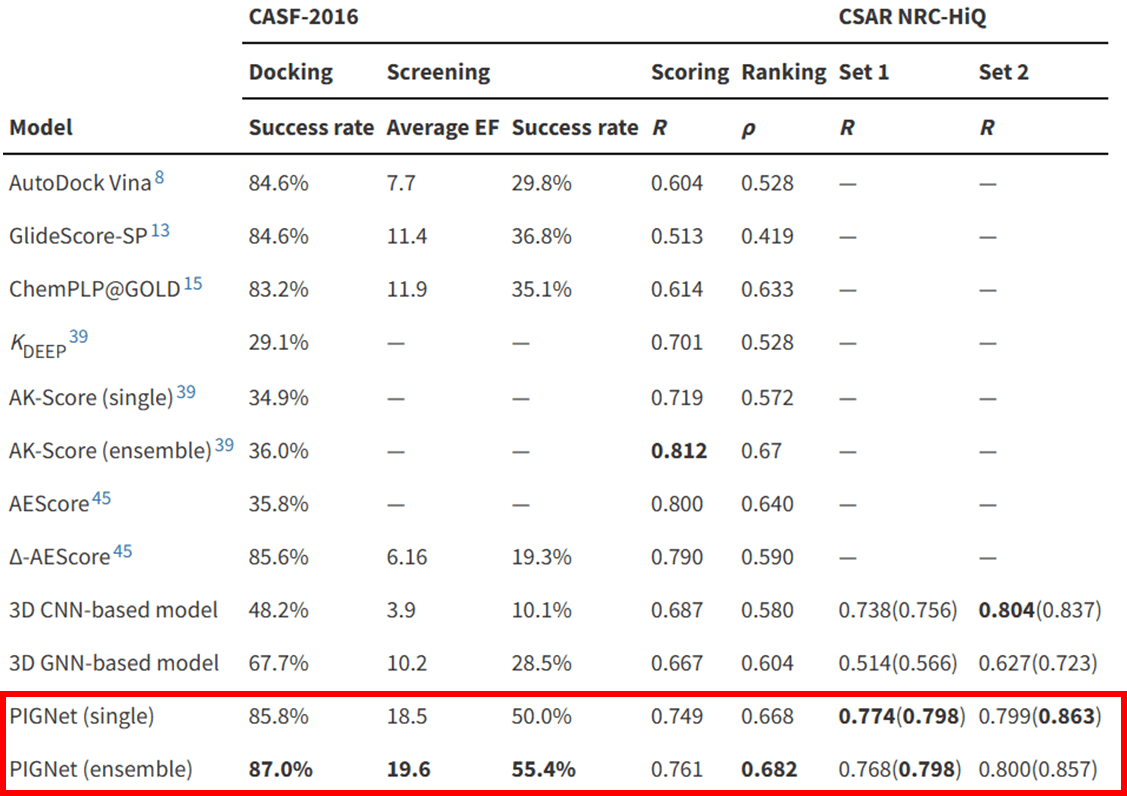
실제로 저희 Hyper Lab 플랫폼에 내장된 binding affinity 예측 AI 모듈인 PIGNet은 binding affinity 예측 벤치마크인 CASF-2016 scoring에서 Pearson 상관 계수 기준 약 0.77의 성능을 보여 줍니다. 즉, Hyper Lab을 통해 다량의 약물에 대한 실험값과 유사한 binding affinity 예측값을 빠른 시간 안에 얻을 수 있으며, 이를 IC50 예측을 위한 feature로 활용할 수 있습니다. 실제로 이번 경진대회에서도 Hyper Lab에서 얻은 binding affinity 예측값을 feature로 추가함으로써 리더보드 순위가 다시 한번 크게 상승하는 성과를 얻었습니다.
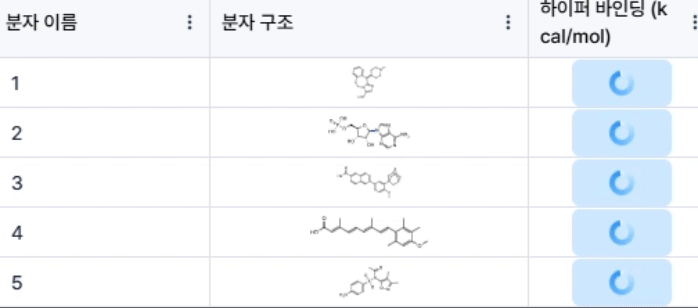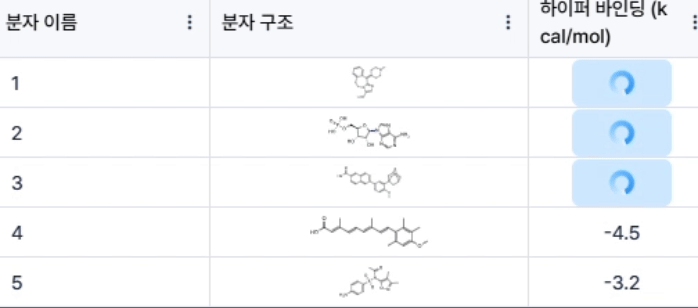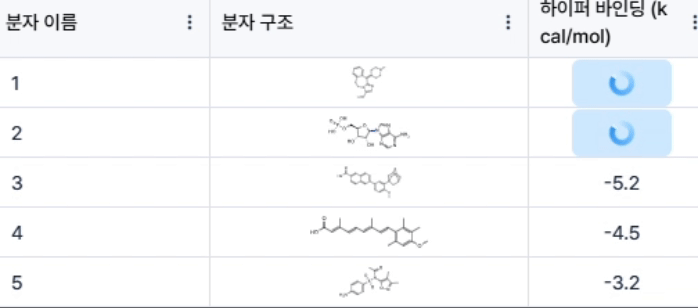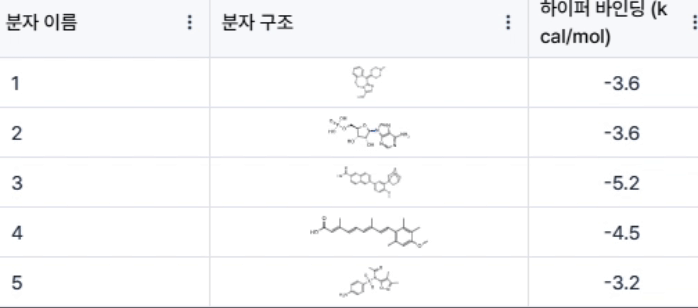
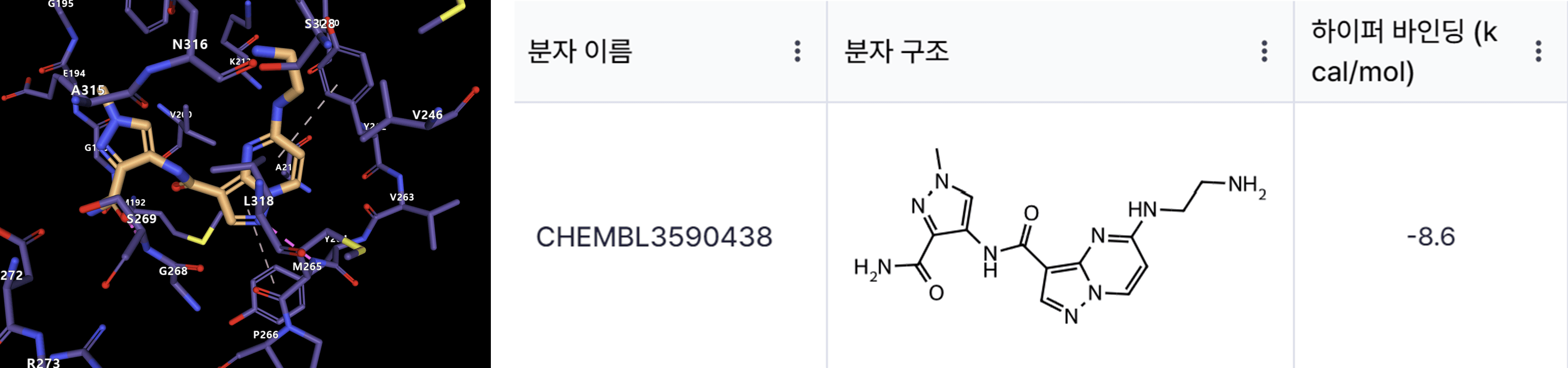
히츠와 함께 도전과 성장을 이어가겠습니다
지금까지 제가 이번 경진대회에서 활용한 여러 AI 신약 개발 기술들에 대해 소개하였습니다. 작년 경진대회에서는 첫 프로젝트로 참여하다 보니 팀원들의 도움이 컸지만, 올해는 지난 1년 동안 쌓아온 경험과 노하우를 바탕으로 스스로 입상하게 되어 더욱 뿌듯했습니다.

또한 훈련 데이터의 한계(데이터 부족과 라벨 분포의 불균형)와 태스크에 대한 이해(IC50의 개념과 binding affinity와의 관계)를 정확히 파악하고, 이를 해결할 수 있는 가설을 수립한 뒤 실험을 진행하여 실제로 리더보드에서 점프업을 경험한 것은 연구자로서 매우 뜻깊은 경험이었습니다. 뿐만 아니라, 일반적으로 실험을 통해서만 얻을 수 있어 AI 예측을 위한 feature로 활용하기 어려운 binding affinity를 Hyper Lab을 통해 예측할 수 있다는 점에서, 저희 히츠의 기술력에도 큰 자부심을 느낄 수 있었습니다. 저의 도전과 성장은 여기서 멈추지 않을 것이며, 앞으로도 히츠와 함께 세상을 더 나은 방향으로 이끄는 데 최선을 다하겠습니다.